다음과 같은 데이터가 있다고 가정해봅시다.
만성신질환 (Chronic kidney disease, CKD) 여부에 따라 급성신손상(Acute kidney injury, AKI)의 단계별로 ICU 입원기간 1일부터 7일 간 Cr의 수치를 비교하는 그래프를 그린다고 가정해봅시다.
우선 CKD 여부에 따른 그래프를 각각 그릴 수 있을 것입니다. 이 때, patchwork 패키지를 이용해 두 개의 그래프를 붙여줄 수 있습니다.
plot_non_ckd <- data[Chronic_kidney_ds==0]|>
ggplot(aes(x=Day, y=mean,
group = as.factor(AKI_stage)))+
geom_line(aes(color=as.factor(AKI_stage))) +
geom_ribbon(aes(y=mean, ymin=mean-sd ,ymax = mean+sd, fill=as.factor(AKI_stage)),
alpha=.2) +
geom_point(aes(color=as.factor(AKI_stage))) +
scale_y_continuous(expand=c(0,0),
limits=c(0,8),
breaks=seq(0,8,1))+
scale_color_discrete(name="AKI Stage") +
scale_fill_discrete(name="AKI Stage") +
theme_classic() +
labs(x="ICU Days",
y= "Average Cr (mg/dL)") +
theme(legend.position = "top")
plot_ckd <- data[Chronic_kidney_ds==1]|>
ggplot(aes(x=Day, y=mean,
group = as.factor(AKI_stage)))+
geom_line(aes(color=as.factor(AKI_stage))) +
geom_ribbon(aes(y=mean, ymin=mean-sd ,ymax = mean+sd, fill=as.factor(AKI_stage)),
alpha=.2) +
geom_point(aes(color=as.factor(AKI_stage))) +
scale_y_continuous(expand=c(0,0),
limits=c(0,8),
breaks=seq(0,8,1))+
scale_color_discrete(name="AKI Stage") +
scale_fill_discrete(name="AKI Stage") +
theme_classic() +
labs(x="ICU Days",
y= "Average Cr (mg/dL)") +
theme(legend.position = "top")
plot_non_ckd + plot_ckd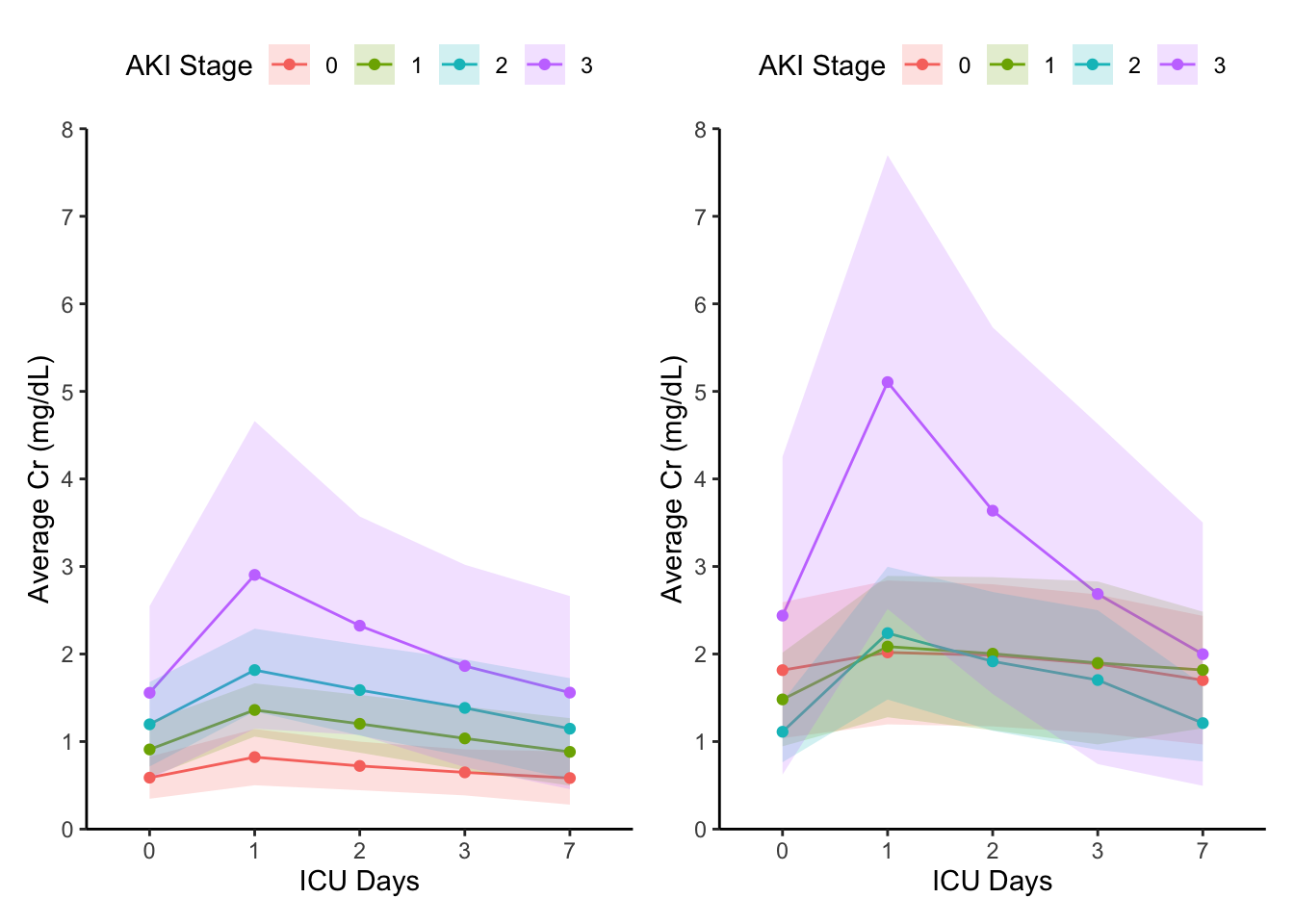
물론 두 개의 그래프를 각각 보여주는 것도 나쁘지 않습니다. 다만 y축이나 legend는 공통된 값들이기 때문에 두 번 보여줄 필요는 없습니다.
그럴 때는 facet_grid() 를 이용해주면 됩니다.
plot <- data|>
ggplot(aes(x=Day, y=mean,
group = as.factor(AKI_stage)))+
geom_line(aes(color=as.factor(AKI_stage))) +
geom_ribbon(aes(y=mean, ymin=mean-sd ,ymax = mean+sd, fill=as.factor(AKI_stage)),
alpha=.2) +
geom_point(aes(color=as.factor(AKI_stage))) +
scale_y_continuous(expand=c(0,0),
limits=c(0,8),
breaks=seq(0,8,1))+
scale_color_discrete(name="AKI Stage") +
scale_fill_discrete(name="AKI Stage") +
theme_classic() +
labs(x="ICU Days",
y= "Average Cr (mg/dL)") +
theme(legend.position = "top")
plot + facet_grid(~Chronic_kidney_ds)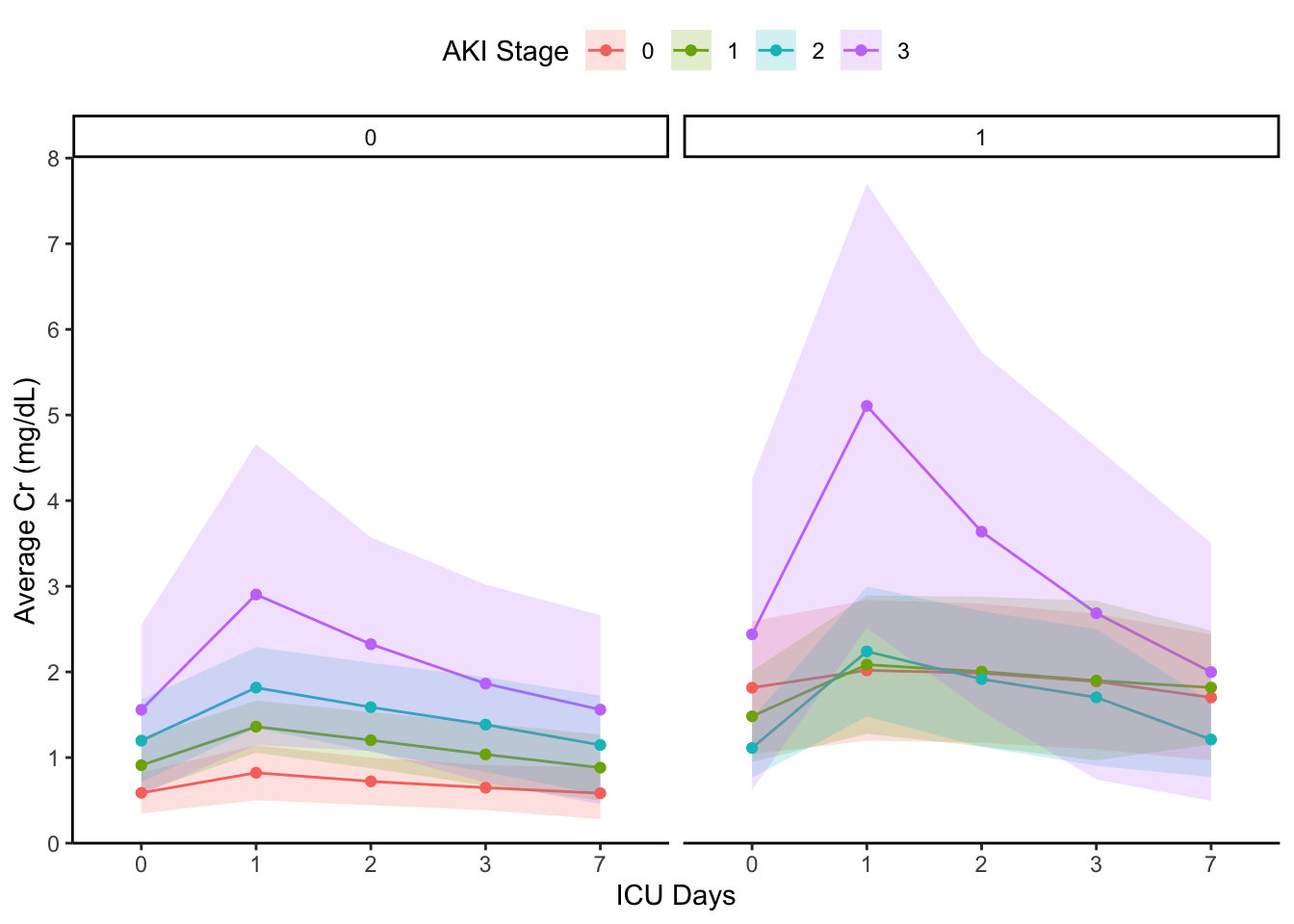
여기서 CKD 변수의 코딩이 0과 1로 되어있어, 그래프에도 0과 1로 나타났습니다. facet의 label을 수정하려면 아래와 같이 labeller를 추가해주면 됩니다.
ckd_label <- c("Non-CKD","CKD")
names(ckd_label) <- c(0,1)
plot +
facet_grid(~Chronic_kidney_ds,
labeller = labeller(Chronic_kidney_ds=ckd_label))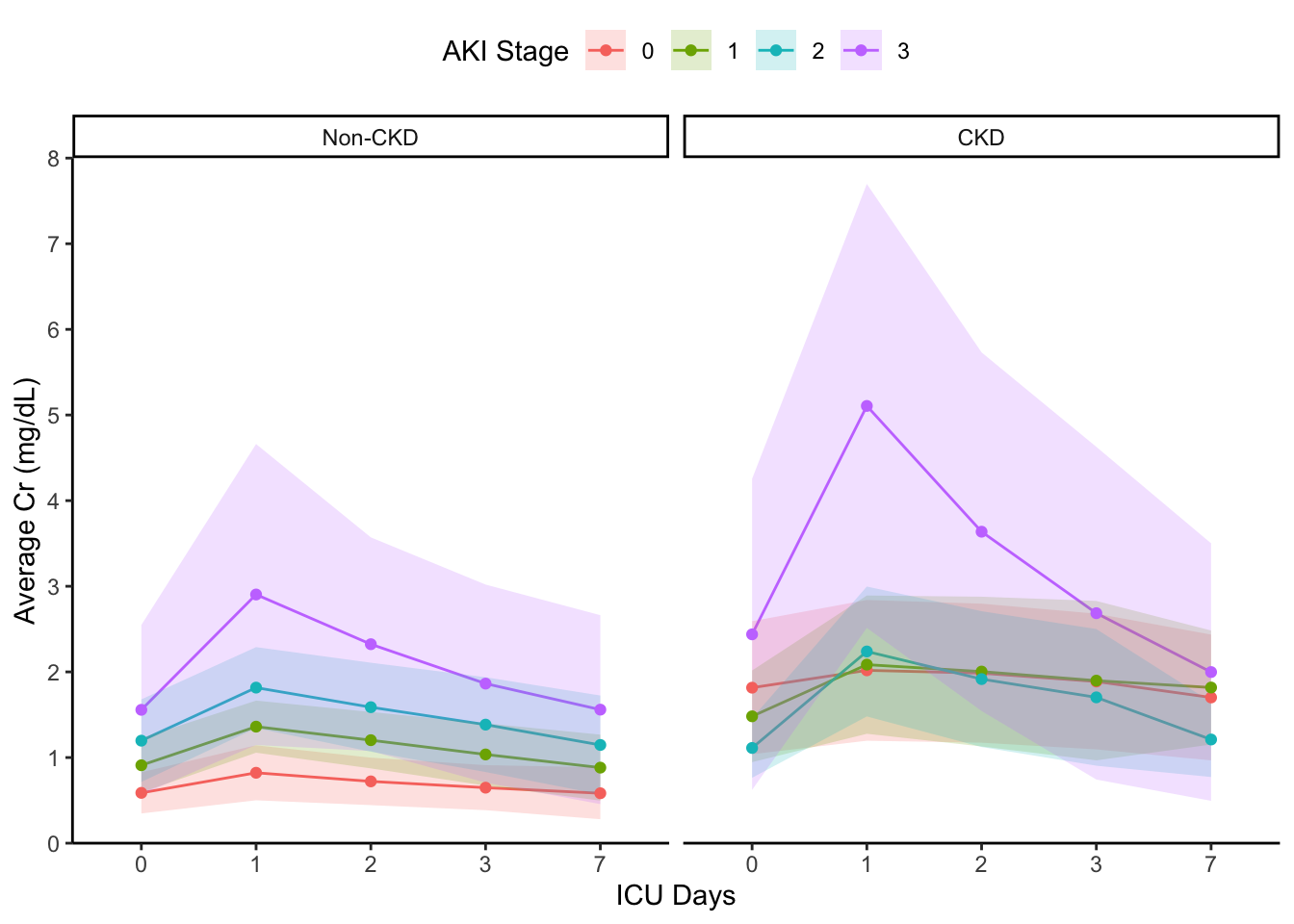
labeller 사용이 어렵다면 데이터의 자체를 리코딩(변경)해주면 됩니다.